【佳学基因检测】癌症序列变异解释与报告标准指南:如何保证肿瘤基因检测的准确性?
ASCO和CAP联合推荐
肺癌基因检测单位选择标准简介
肺癌是中国第一大发病率癌症,佳学基因投入大量人力物力专注于良性、恶性肿瘤的产生原因、阻抑方式、靶向药物及免疫药物的选择和使用方法。提出基因解码是肿瘤早期预测、个性化根治的信息依据,并指出基因解码解密基因序列变异与肿瘤诊断与治疗的关系要优先于基于数据库比对的基因检测。关于癌症序列变异解释与报告标准指导是将整个行业推向正确方向的第一步。
1. 引言
基于二代测序技术的肿瘤检测已越来越多的应用于临床实验室中,但目前在不同实验室间存在检测方法、报告内容等方面的差异,这对遗传检测的解读以及普及应用造成了一定的影响。因此,在不同实验室间建立统一的分子检测结果的解读和报告标准,及建立行业标准,显得尤为迫切。
在2015年春天,在美国专门成立了一个以临床实验室为核心的工作组,其组成包含了分子病理协会(AMP)、美国医学遗传学与基因组学协会(ACMG)、美国临床肿瘤学会(ASCO)与美国病理学家协会(CAP)的一线专家,该工作组的主要工作为对肿瘤及疑似肿瘤相关的序列变异检测建立检测标准并在行业达成共识。
该工作组首先对北美地区超过40家的临床检测实验室进行了问卷调查,结果显示不同实验室在检测组织类型、检测基因数量、是否检测肿瘤组织全外显子组或全基因组、以及其他细节方面都存在较大差别,此外在不同单位的检测报告的报告内容方面也存在较多差异。该工作组认为,为医疗机构提供分类的遗传变异报告对病人及整个医疗行业都极为重要,包括:提供精确的肿瘤对靶向治疗反应性信息;建立国家级别的医疗指南;以及与临床试验合作,对建立不同实验室间的通用标准提供支持。基于以上这些考量,工作组专家们根据已有数据、文献报道和专业知识,给出以下指南建议。
图1 AMP对NGS技术及NGS结果解读的调研
图注:
A: MAF阈值;B: 变异分类数目;C:报告中是否包含治疗性建议;D: 报告中是否包含潜在的生殖细胞突变;
E: 报告是否包含变异等位频率Variant allele frequency (VAF);F: 报告是否包含基因组坐标;
G: 报告是否包含转录本ID(Transcript accession);H: 报告是否包含不符合质控的基因/区间。
2. 术语
dbSNP, The Database of Short Genetic Variation;
ExAC, Exome Aggregation Consortium;
IARC, International Agency for Research on Cancer;
NCBI, National Center for Biotechnology Information;
SNV, single-nucleotide variant;
UCSC, University of California, Santa Cruz;
WHO, World Health Organization;
FDA, Food and Drug Administration;
CNV, copy number variation;
COSMIC, Catalog of Somatic Mutations in Cancer;
HGMD, Human Gene Mutation Database;
indel, insertion and deletion;
LOF, loss of function;
MAF, minor allele frequency;
PG, professional guideline;
TCGA, The Cancer Genome Atlas;
VAF, variant allele frequency.
3. 数据库
3.1 基因组数据库
3.1.1 大型肿瘤基因组数据库
1. The National Cancer Institute’s Genome Data Commons:
- The Cancer Genome Atlas(TCGA)
- Therapeutically Applicable Research to Generate Effective Therapies(TARGET)
- the Cancer Genome Characterization Initiative
2. Catalog of Somatic Mutations in Cancer
3.1.2 体细胞序列变异解释相关数据库
表1 体细胞序列变异解释相关数据库
| Utility/function | Database | Location (web address) |
| Population databases to exclude polymorphisms | √1000 Genomes Project16√ | http://browser.1000genomes.org |
| Exome Variant Server | http://evs.gs.washington.edu/EVS | |
| √dbSNP17√ | http://www.ncbi.nlm.nih.gov/snp | |
| dbVar18√ | http://www.ncbi.nlm.nih.gov/dbvar | |
| ExAC√ | http://exac.broadinstitute.org | |
| Cancer-specific variant databases | √Catalog of Somatic Mutations in Cancer19√ | http://cancer.sanger.ac.uk/cosmic |
| My Cancer Genome | http://www.mycancergenome.org | |
| Personalized cancer therapy, MD Anderson Cancer Center | https://pct.mdanderson.org | |
| cBioPortal, Memorial Sloan Kettering Cancer Center20 | http://www.cbioportal.org | |
| Intogen21 | https://www.intogen.org/search | |
| ClinicalTrials.gov | https://clinicaltrials.gov | |
| IARC (WHO) TP53 mutation database22 | http://p53.iarc.fr | |
| Pediatric Cancer Genome Project (St. Jude Children’s Research Hospital–Washington University) | http://explorepcgp.org | |
| International Cancer Genome Consortium23√ | https://dcc.icgc.org | |
| Sequence repositories and data hosts | NCBI Genome | http://www.ncbi.nlm.nih.gov/genome |
| RefSeqGene24 | http://www.ncbi.nlm.nih.gov/refseq/rsg | |
| Locus Reference Genomic25 | http://www.lrg-sequence.org | |
| UCSC table browser26 | https://genome.ucsc.edu/cgi-bin/hgTables | |
| Ensemble BioMart27√ | http://useast.ensembl.org/biomart/martview | |
| Other disease/mutation databases useful in the context of variant interpretation for cancer genomics | √ClinVar28√ | http://www.ncbi.nlm.nih.gov/clinvar |
| √Human Gene Mutation Database29√ | http://www.hgmd.org | |
| Leiden Open Variation Database30 | http://www.lovd.nl | |
| dbNSFP (compiled database of precomputed in silico prediction scores for nonsynonymous SNVs)31√ | https://sites.google.com/site/jpopgen/dbNSFP | |
| Ensemble Variant Effect Predictor15 | http://www.ensembl.org/info/docs/tools/vep/index.html |
表注:
表格为不完全统计,最后更新日期2016年6月7日。
3.1.3 临床实验室在应用公共数据库时一般的注意事项
1. 了解数据库内容及数据组织形式;
2. 特别注意数据库的限制,避免检测结果过度解读;
3. 注意基因组与转录组的组装版本,应与Human Genome Variation Society (HGVS)注释一致;
4. 根据已有资源评估基因组测序质量;
5. 确认用于病理诊断的数据质量。
3.2 参考序列数据库
常见参考序列数据库用于人类基因组组装及相关信息,包括基因组坐标,转录本编号与版本,外显子边界等等。常见的有:
- √RefSeq (National Center for Biotechnology Information Reference Sequence Database,https://www.ncbi.nlm.nih.gov/refseq, last accessed January 2, 2016),
- √Ensembl (http://www.ensembl.org/index.html, last accessed January 2, 2016),
- Locus Reference Genomic (https://www.lrg-sequence.org,last accessed February 2, 2016)
3.3 人群数据库
人群数据库提供基因多态性、人群等位基因频率等信息,用于区分测序结果中的多态性与突变,常见的有:
- The Database of Short Genetic Variation (the National Center for Biotechnology Information database of genetic variation)
- Exome Variant Server
- √Exome Aggregation Consortium
3.4 肿瘤特异性数据库
肿瘤特异性数据库提供汇总之后的不同肿瘤类型及亚型间的序列变异发生率,还包括发表的文献、临床试验、成果数据等数据,见表1 Cancer-specific variant databases。
3.5 原发性变异数据库
原发性变异数据库(Constitutional Variant Databases)主要用于区分生殖系变异与体细胞变异,常用的有:
- √Human Gene Mutation Database
- √ClinVar (http://www.ncbi.nlm.nih.gov/clinvar)
3.6 内部数据库(实验室产生)
临床实验室根据内部资源独立建立的数据库,可用于变异注释,指南鼓励将这些体细胞变异的注释数据逐步整合到公共数据库资源中去促进体细胞变异的解释。
4. 生物信息学预测算法
生物信息学预测算法常用于预测基因中核苷酸的变化是否会影响基因的结构和蛋白质的功能,总体上看,目前的生物信息学算法还无法保证足够高的特异性(大约60%-80%),因此建议不要将预测结果作为临床分类与临床决策的证据。
表2 预测序列突变对基因结构和蛋白质功能影响的算法
| Utilsity/function | Algorithm/software | Location (web address) |
| Missense SNV | √PolyPhen237√ | http://genetics.bwh.harvard.edu/pph2 |
| √SIFT38√ | http://sift.jcvi.org | |
| MutationAssessor39√ | http://mutationassessor.org | |
| MutationTaster41√ | http://www.mutationtaster.org | |
| PROVEAN45 | http://provean.jcvi.org/index.php | |
| Condel46 | http://bg.upf.edu/blog/2012/12/condel-for-prioritization-of-variants-involved-in-hereditary-diseases-and-transfic-for-cancer | |
| CoVEC40 | https://sourceforge.net/projects/covec/files | |
| CADD47√ | http://cadd.gs.washington.edu | |
| GERP++48√ | http://mendel.stanford.edu/sidowlab/downloads/gerp/index.html | |
| PhyloP and PhastCons49√ | http://compgen.bscb.cornell.edu/phast | |
| Splice site prediction | Human Splicing Finder42 | http://www.umd.be/HSF3 |
| MaxEntScan43 | http://genes.mit.edu/burgelab/maxent/Xmaxentscan_scoreseq.html | |
| NetGene244 | http://www.cbs.dtu.dk/services/NetGene2 | |
| NNSplice50 | http://www.fruitfly.org/seq_tools/splice.html | |
| GeneSplicer51 | http://www.cbcb.umd.edu/software/GeneSplicer/gene_spl.shtml |
表注:
表格为不完全统计,最后更新日期2016年6月7日。
5. 变异鉴定与注释
通常由软件来鉴定和注释变异,比如点突变、插入缺失、结构变异和拷贝数变异。临床实验室需注意软件工具的局限性。测序深度、等位基因频率等信息需加以考虑,在肿瘤异质性评估中等位基因频率对体细胞突变的解读尤为重要,特别是在缺乏配对正常样本的时候。目前较为通行的注释文件格式为VCF格式,可从中分析相关信息。常见软件有:
表3 突变注释软件
| Software | Location (URL) |
| Annovar√ | http://annovar.openbioinformatics.org/en/latest/ |
| snpEff | http://snpeff.sourceforge.net/ |
| SeattleSeq | http://snp.gs.washington.edu/SeattleSeqAnnotation138/ |
| AnnTools | http://anntools.sourceforge.net/ |
| NGS-SNP | https://www.ualberta.ca/~stothard/downloads/NGS-SNP/ |
| VEP (Variant Effect Predictor) | http://useast.ensembl.org/info/docs/tools/vep/index.html |
表注:
表格为不完全统计,最后更新日期2016年6月7日。
表4 突变解析软件
| Variant caller | Location (URL) |
| MuTect v1.1.555 | https://www.broadinstitute.org/cancer/cga/mutect |
| GATK – MuTect v2 | https://www.broadinstitute.org/gatk/guide/tooldocs/org_broadinstitute_gatk_tools_walkers_cancer_m2_MuTect2.php |
| VarScan 256 | http://dkoboldt.github.io/varscan/ |
| VarDict57 | https://github.com/AstraZeneca-NGS/VarDict |
| Sterlka58 | https://sites.google.com/site/strelkasomaticvariantcaller/ |
| FreeBayes59 | https://github.com/ekg/freebayes |
| Scalpel60 | http://scalpel.sourceforge.net/ |
| Pindel61 | http://gmt.genome.wustl.edu/packages/pindel/ |
| SAMtools62 | http://samtools.sourceforge.net/ |
| Torrent Suite Variant Caller | https://github.com/iontorrent/TS |
| SomaticSniper63 | http://gmt.genome.wustl.edu/packages/somatic-sniper/ |
表注:
表格为不完全统计,最后更新日期2016年6月7日。
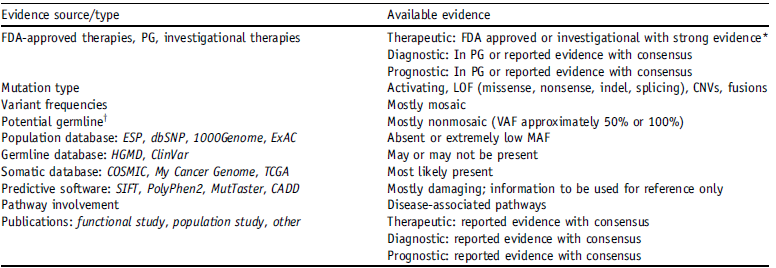
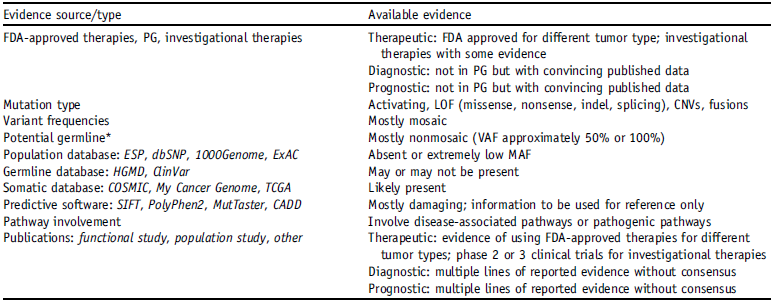
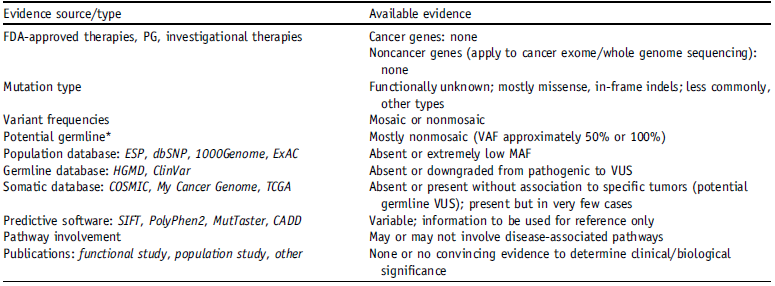
6. 基于证据的体细胞变异分类的指导意见
基于循证医学的体细胞变异分类的指导意见(Proposed Guideline for Evidence-Based Categorization of Somatic Variants),根据文献报道及工作组专家共识,提议将基因检测相关临床及实验证据分为4级:
表5 临床及实验证据分类
| Category | Therapeutic | Diagnosis | Prognosis |
| Level A | Biomarkers that predict response or resistance to FDA-approved therapies for a specific type of tumor | Biomarkers included in professional guidelines as diagnostic for a specific type of tumor | Biomarkers included in professional guidelines as prognostic for a specific type of tumor |
| Biomarkers included in professional guidelines that predict response or resistance to therapies for a specific type of tumor | |||
| Level B | Biomarkers that predict response or resistance to therapies for a specific type of tumor based on well-powered studies with consensus from experts in the field | Biomarkers of diagnostic significance for a specific type of tumor based on well-powered studies with consensus from experts in the field | Biomarkers of prognostic significance for a specific type of tumor based on well-powered studies with consensus from experts in the field |
| Level C | Biomarkers that predict response or resistance to therapies approved by the FDA or professional societies for a different type of tumor | Biomarkers of diagnostic significance based on the results of multiple small studies | Biomarkers of prognostic significance based on the results of multiple small studies |
| Biomarkers that serve as inclusion criteria for clinical trials | |||
| Level D | Biomarkers of prognostic significance based on the results of multiple small studies | Biomarkers that may assist disease diagnosis themselves or along with other biomarkers based on small studies or a few case reports | Biomarkers that may assist disease prognosis themselves or along with other biomarkers based on small studies or a few case reports |
表注:
A级:可作为FDA批准治疗某种特异性肿瘤的生物标志物(biomarker),或被收录于某些特殊类型肿瘤的诊断、治疗、预后评价的权威指南;
B级:可作为基于强力临床试验证据和专家共识的治疗某种特异性肿瘤的生物标志物,或用于诊断、预后评估;
C级:可用于指导FDA批准的标注外用药,或基于多个小规模研究的证据的诊断/预后评估;
D级:结果不确定的临床前研究,可作为辅助标志物,基于小规模研究且未达成共识。
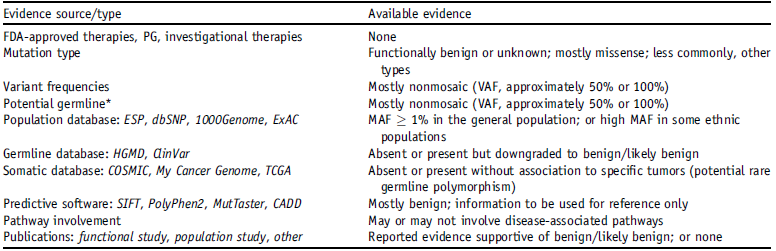
图2 基于证据体细胞变异分类(临床影响力)
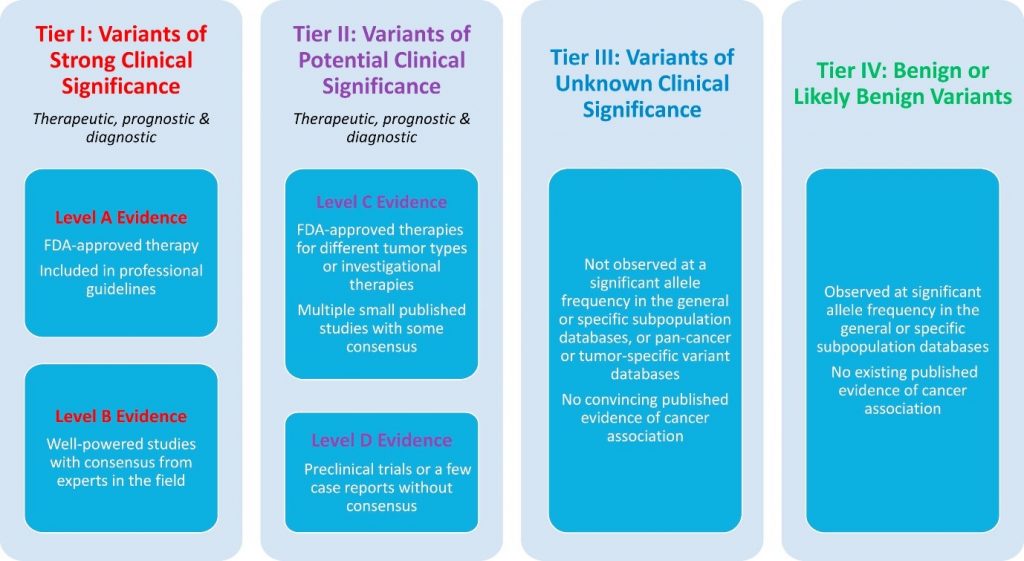
图注:
I级:具有强临床意义的变异(A级和B级证据);
II级:具有潜在临床意义的变异(C级或D级证据);
III级:变异的临床意义不确定;
IV级:与良性或疑似良性相关的变异。
7. 检测结果报告与解读
- 所有检测到的变异都需要根据临床影响力4级分级系统进行分级标识;
- 所有检测到的变异都需要按照HUGO基因命名委员会规则注释与报告,报告中也应包含其他必要信息;
- 生殖系变异在报告中可不加以区分,若报告生殖系变异需符合当地法律与政策(患者需签署知情同意书等);
- 报告检测到变异的临床意义;
- 报告可随相关医学知识的更新而发生变化,当有特殊需求时可考虑更新报告;
- 实验方法应随检测结果一同报告;
- 可将检测报告与电子病历系统整合。
注:
1. NGS分析无法区分体系变异与生殖系变异;
2. 生殖系变异的报告需要依据ACMG/AMP指南。
8. 结论
在癌症基因组谱中越来越多地使用NGS技术,给临床实验室带来了新的挑战。分子专业人员的关键任务之一是,基因测序检测到的癌症相关序列变体的解释和报告的标准化。拟议的“癌症序列变异解释与报告标准指南”代表了工作组成员的专家一致意见及他们代表的利益相关者的意见。该建议描述了一个基于证据的变量分类系统,以及变量注释,分类和报告的过程。该报告还列出了常用于NGS数据分析的有用的生物信息学工具和数据库。这些建议应作为临床实验室专业人员和肿瘤学家的教育资源,以辅助变体解释和临床决策。我们希望本文提供的指南将在癌症基因组学领域得到广泛应用,并推动癌症患者的基因组检测和精确治疗实践。
9. 免责声明
分子病理学协会(AMP)临床实践指南和报告的开发,旨在为实践和特定领域的实践提供指导和建议,帮助实验室和其他卫生保健专业人员。指南或报告不应被视为包括所有正确的方式或方法,或排除其他方式或方法。指南或报告不能保证任何具体结果,也不能建立一种护理标准。指南或报告不是要指定对特定患者的治疗。治疗决定必须基于健康护理提供者和每个患者的个体情况的独立判断。 AMP对本指南或报告不作任何明示或暗示的保证,特别不包括任何对特定用途或目的适销性或适用性的保证。 AMP对与使用本文所含信息相关的直接,间接,特殊,偶然或继发性损失概不负责。
10. 参考资料
http://jmd.amjpathol.org/article/S1525-1578(16)30223-9/fulltext#appsec1
(责任编辑:admin)



